
揭秘共定位分析:使用Fiji进行精准的蛋白质共定位研究
一站式指南:轻松掌握最强大的科学图像共定位分析工具及其关键技术
共定位分析核心要点
- Fiji不仅仅是ImageJ的增强版本,它提供了专门的共定位插件如Coloc 2和JACoP,能够精确量化不同荧光通道之间的空间重叠
- 成功的共定位分析依赖于良好的图像质量和正确的图像预处理,包括背景校正、通道分离和适当的阈值设置
- 不同的共定位分析方法(如Pearson相关系数、Manders重叠系数等)适用于不同类型的生物学问题,理解它们的区别至关重要
Fiji共定位分析基础概念
Fiji(全称"Fiji Is Just ImageJ")是一个功能强大的开源图像分析软件,特别适合生物医学领域的图像处理。共定位分析(Colocalization Analysis)是研究两种或多种荧光标记蛋白质或分子在细胞或组织中空间分布重叠程度的重要技术。理解共定位对于研究蛋白质相互作用、信号传导和细胞功能至关重要。
在开始共定位分析前,需要了解几个基本概念:
- 共定位(Colocalization):两种或多种荧光标记在显微镜分辨率范围内出现在同一位置
- 相关性(Correlation):两个通道的荧光强度变化是否呈正相关、负相关或无相关
- 共存(Co-occurrence):两种标记是否仅仅存在于同一位置,而不考虑强度关系
Fiji安装与准备工作
在进行共定位分析前,首先需要安装Fiji软件:
- 访问Fiji官方网站(https://fiji.sc/)下载最新版本
- 按照安装指南完成安装(Fiji通常包含多数必要的插件)
- 确保Fiji已安装Coloc 2或JACoP插件(在"Plugins"菜单中查看)
- 准备高质量的多通道荧光图像(通常包含至少两个不同颜色通道)
图像采集原则与质量控制
高质量的共定位分析始于高质量的图像采集。请确保:
- 使用适当的显微镜设置,避免通道间的串扰(bleed-through)
- 图像分辨率足够高,能够区分所研究的结构
- 避免像素饱和,保持图像动态范围
- 在采集过程中,样本不发生明显的光漂白或移动
- 使用合适的对照样本验证共定位结果
使用Fiji进行共定位分析的步骤
图像预处理
在进行共定位分析前,适当的图像预处理可以显著提高结果的可靠性:
- 打开多通道图像:使用"File > Open"打开您的多通道图像
- 分离通道:使用"Image > Color > Split Channels"将图像分离为单独的通道
- 背景校正:
- 使用"Process > Subtract Background"去除背景噪声
- 或使用ROI(感兴趣区域)在无信号区域测量背景值,然后手动减去
- 降噪处理:如需要,使用"Process > Filters > Gaussian Blur"或中值滤波减少噪声
- 阈值设置:使用"Image > Adjust > Threshold"设置适当的阈值,排除背景像素
使用Coloc 2插件进行分析
Coloc 2是Fiji中最强大的共定位分析插件之一,提供多种统计方法:
- 打开Coloc 2:选择"Plugins > Colocalization > Coloc 2"
- 选择通道:在弹出窗口中,选择需要分析的两个通道图像
- 设置ROI(可选):如果只需分析图像的特定区域,可以使用ROI管理器设置
- 选择分析选项:
- 勾选所需的分析方法(如Pearson、Manders、Li等系数)
- 设置阈值方法(如Costes自动阈值)
- 选择是否进行统计显著性检验(如Costes randomization)
- 运行分析:点击"OK"按钮开始分析
- 查看结果:分析完成后,Coloc 2会显示多个结果窗口,包括散点图、系数值和统计结果
替代方法:使用JACoP插件
JACoP(Just Another Colocalization Plugin)是另一个常用的共定位分析插件:
- 打开JACoP:选择"Plugins > Analysis > JACoP"
- 选择通道:选择需要分析的两个通道图像
- 选择分析选项:JACoP提供多种共定位分析方法,包括Pearson、Manders、对象计数等
- 运行分析:点击"Run"按钮开始分析
- 查看结果:JACoP会显示一个结果窗口,包含所有计算的系数和统计结果
上图展示了Fiji中不同共定位分析插件的功能对比,Coloc 2和JACoP是最常用的两个插件,但根据具体需求,可能需要选择不同的工具。
理解共定位系数和参数
| 系数名称 | 值范围 | 适用场景 | 优势 | 局限性 |
|---|---|---|---|---|
| Pearson相关系数(PCC) | -1至+1 | 评估两个通道的整体线性相关性 | 不受背景和信号强度影响 | 无法区分阴性共定位和无共定位 |
| Manders重叠系数(MOC) | 0至1 | 测量通道间的重叠程度 | 容易理解,可提供每个通道的分数 | 受背景信号影响大 |
| Li强度相关商(ICQ) | -0.5至+0.5 | 适用于非线性相关关系 | 对非线性关系敏感 | 解释复杂 |
| Costes自动阈值 | 阈值值 | 自动确定背景阈值 | 减少主观因素,提高一致性 | 在某些低信噪比图像中可能不准确 |
| 对象基共定位 | 对象数量/百分比 | 分析离散结构的共定位 | 适合颗粒和点状结构分析 | 需要先进行对象分割 |
共定位分析进阶技术
3D和多维共定位分析
对于Z-stack或多维数据集,Fiji提供了专门的3D共定位分析工具:
- 对于Z-stack图像,可使用"Image > Stacks > Z Project"将3D数据转换为2D进行分析
- 或使用3D Object Counter和DiAna等插件直接在3D环境中分析共定位
- 时间序列数据可使用"Image > Hyperstacks"工具组织和分析
结果解释与验证
共定位分析的结果解释需要谨慎:
- 对照验证:使用阳性和阴性对照验证结果的可靠性
- 生物学解释:将数值与生物学背景结合解释
- 统计分析:使用多个样本进行统计比较
- 结果可视化:使用伪彩图(以白色或黄色显示共定位区域)直观展示共定位
共定位伪像的识别与避免
共定位分析中的伪像可能导致错误结论,需要注意:
- 荧光通道间的串扰(bleed-through):使用单标记对照检查
- 显微镜分辨率限制:考虑超分辨显微技术
- 样本移动或漂移:使用图像配准技术校正
- 高背景噪声:适当的背景校正和阈值设置
上图展示了使用Fiji进行共定位分析的完整工作流程,从准备工作到结果展示的每个环节都需要认真对待,以确保分析结果的可靠性。
共定位图像实例与分析
以下是几个典型的共定位分析图像示例,展示了不同程度的共定位情况:
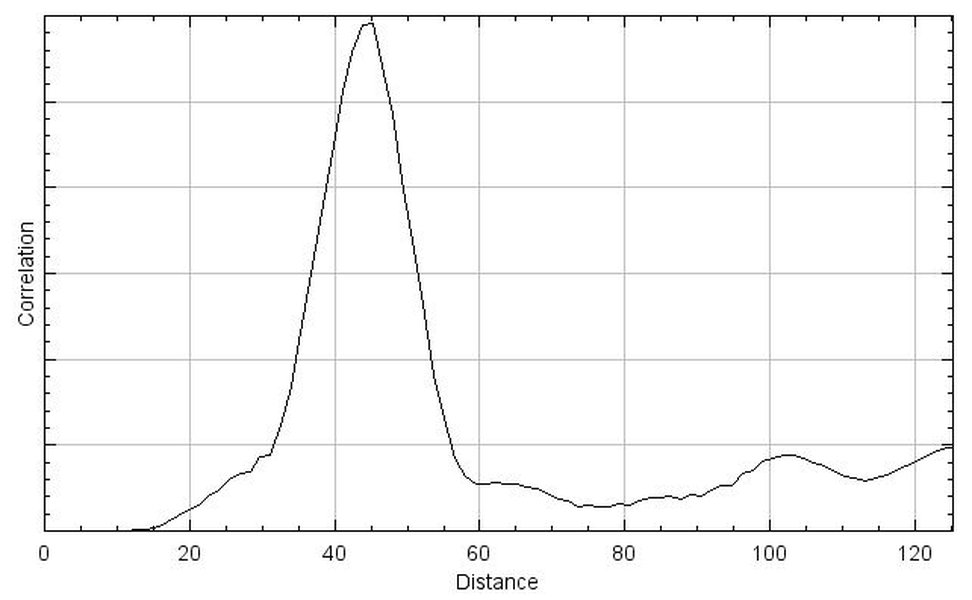
图:ImageJ/Fiji中的共定位分析曲线示例,展示了在不同距离或时间点的共定位相关性变化
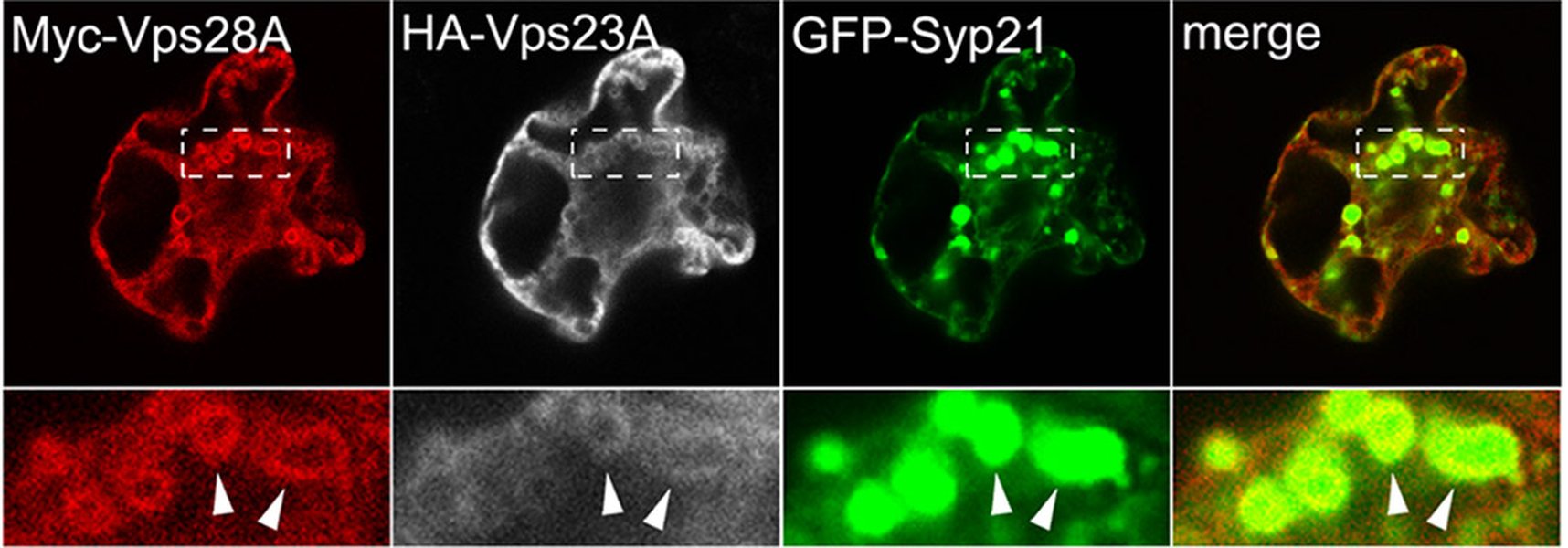
图:荧光显微镜图像的共定位分析,黄色区域表示红色和绿色通道重叠的共定位区域
共定位分析视频教程
以下视频详细展示了如何使用Fiji进行共定位分析的操作步骤,包括插件的使用、参数设置和结果解释:
这个视频展示了如何使用FIJI (ImageJ)的JACoP BIOP版本测量荧光显微镜图像中的共定位、共存和相关性,内容详实,适合初学者学习。
常见问题与解答
参考资料
- Coloc 2 - ImageJ Wiki
- Colocalization Analysis - ImageJ Wiki
- Fiji Colocalisation Analysis GitHub Repository
- How to choose the best colocalization method - LinkedIn
- How to perform colocalization analysis in 3D/4D images - LinkedIn
- Validating and comparing results of different colocalization methods - LinkedIn
- The Principles of Colocalization Microscopy and Image Analysis - Kolaido
推荐阅读
Last updated April 9, 2025